Рис. 7.10(epig). Модель сканирования транскриптома у инфузорий
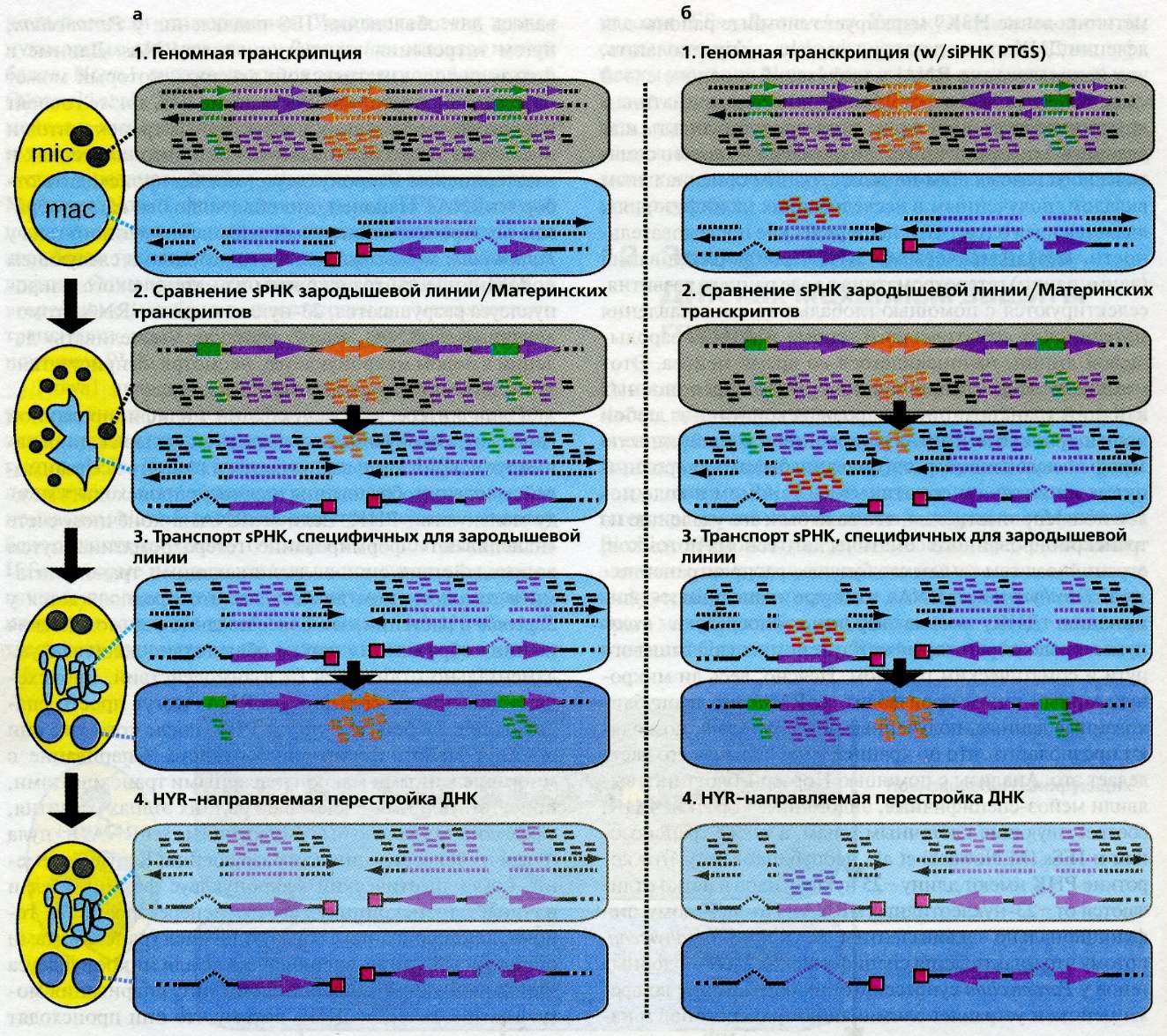
Модель сканирования транскриптома для сравнения соматического генома и генома зародышевой линии.
(а) Регулирование "по умолчанию".
(1) После начала мейоза большинство или даже все макронуклеарные и микронуклеарные геномы транскрибируются; штриховые линии в макронуклеусе представляют неохарактеризованные транскрипты. В микронуклеусе транскрипция является двунаправленной и приводит к образованию scnRNAs (коротких двунитевых молекул) для последовательностей всех типов (клеточных генов, светлорозовые стрелки; транспозонов, оранжевая двойная стрелка; IES, зеленые прямоугольники). Эти scnRNAs экспортируются в материнский макронуклеус, где они могут спариваться с гомологичными соматическими транскриптами. Спаривание может происходить и в цитоплазме. scnRNAs, спаривающиеся с гомологичными транскриптами, секвестрируются или разрушаются, тогда как scnRNAs, специфичные для микронуклеуса, реэкспортируются в развивающийся зиготический макронуклеус, где они спариваются с гомологичными последовательностями (ДНК или только что образовавшимися транскриптами), "нацеливая" таким образом метилирование по НЗК9 на специфичные для микронуклеуса последовательности (транспозоны и IESs).
(4) Маркированные последовательности элиминируются.
(б) Эффекты посттранскрипционного сайленсинга гена в материнском макронуклеусе.
Экспериментальная индукция посттранскрипционного сайленсинга многокопийными трансгенами или dsRNA приводит к образованию двунитевых siRNAs, гомологичных этому гену (показано красным цветом). Эти siRNAs разрушают гомологичные материнские соматические транскрипты, так что гомологичные scnRNAs не будут инактивированы и смогут "нацеливать" делецию данного гена в развивающемся зиготическом макронуклеусе.